Con questo Jupyter Notebook voglio mostrare come ho elaborato i dati CAMS relativi al PM10 di cui ho parlato in questo articolo. I dati grezzi sono in formato NetCDF e di come gestire questo particolare formato dati ne ho parlato in un altro articolo che, nemmeno a farlo apposta, è incentrato sui dati CAMS.
Prima di iniziare¶
Librerie¶
Le librerie Python usate per raggiungere lo scopo sono state diverse e le vedrete di seguito. Una però deve essere menzionata tra tutte Dask; senza l'uso di questa librerie non mi sarebbe stato possibile velocizzare i processamenti che, nonostante tutto, sono durati ore.
from pathlib import Path
import geopandas as gpd
import matplotlib.pyplot as plt
import xarray as xr
from geocube.api.core import make_geocube
from xrspatial.zonal import stats
import pandas as pd
import dask.dataframe as dd
import seaborn as sns
Fonti dati¶
I dati del PM10 dal 2013 allo scorso marzo sono il perno di questa analisi a cui ho associato i dati vettoriali dell'ISTAT delle province target(quelle che ricadono nelle regioni con codice da 1 a 11). Dopo aver scaricato i dati CAMS li ho preprocessati in modo da avere un file .nc per anno anche per risolvere il problema di cui ho parlato nel workaround qui.
nc_data_path = Path("/home/max/Desktop/pianura_padana/processed/netcdf")
nc_files = list(nc_data_path.glob("*.nc"))
nc_files
[PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2021-forecasts.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2017-reanalyses.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2020-reanalyses.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2014-reanalyses.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2013-reanalyses.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2016-reanalyses.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2023-forecasts.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2019-reanalyses.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2015-reanalyses.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2018-reanalyses.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2022-forecasts.nc'),
PosixPath('/home/max/Desktop/pianura_padana/processed/netcdf/particulate_matter_10um-2024-forecasts.nc')]
target_zones_boundaries = Path("/home/max/Desktop/DEV/DrakoNotebook/open_dataset/Limiti01012023_g/ProvCM01012023_g/ProvCM01012023_g_WGS84.shp")
target_zones = gpd.read_file(target_zones_boundaries)
target_zones = target_zones[target_zones['COD_REG'].isin(list(range(1, 12)))]
# Nel prossimo passaggio effettuto una cambio di EPSG poichè i dati CAMS usano il 4326 mentre quelli ISTAT sono in 32632.
target_zones = target_zones.to_crs(4326).sort_values('DEN_UTS')
target_zones
| COD_RIP | COD_REG | COD_PROV | COD_CM | COD_UTS | DEN_PROV | DEN_CM | DEN_UTS | SIGLA | TIPO_UTS | Shape_Area | geometry | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 5 | 1 | 1 | 6 | 0 | 6 | Alessandria | - | Alessandria | AL | Provincia | 3.560310e+09 | POLYGON ((8.40549 45.20148, 8.41749 45.19846, ... |
| 41 | 3 | 11 | 42 | 0 | 42 | Ancona | - | Ancona | AN | Provincia | 1.961932e+09 | POLYGON ((13.21155 43.72501, 13.21979 43.71770... |
| 6 | 1 | 2 | 7 | 0 | 7 | Aosta | - | Aosta | AO | Provincia | 3.258838e+09 | POLYGON ((7.58857 45.97075, 7.58981 45.97073, ... |
| 50 | 3 | 9 | 51 | 0 | 51 | Arezzo | - | Arezzo | AR | Provincia | 3.233326e+09 | MULTIPOLYGON (((12.21800 43.60394, 12.21816 43... |
| 43 | 3 | 11 | 44 | 0 | 44 | Ascoli Piceno | - | Ascoli Piceno | AP | Provincia | 1.229268e+09 | POLYGON ((13.78663 43.07104, 13.79679 43.06211... |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 26 | 2 | 5 | 27 | 227 | 227 | - | Venezia | Venezia | VE | Citta metropolitana | 2.473732e+09 | POLYGON ((12.79994 45.82573, 12.80158 45.82316... |
| 102 | 1 | 1 | 103 | 0 | 103 | Verbano-Cusio-Ossola | - | Verbano-Cusio-Ossola | VB | Provincia | 2.262145e+09 | POLYGON ((8.44976 46.46176, 8.46176 46.45081, ... |
| 1 | 1 | 1 | 2 | 0 | 2 | Vercelli | - | Vercelli | VC | Provincia | 2.082097e+09 | POLYGON ((8.20447 45.93567, 8.21365 45.92490, ... |
| 22 | 2 | 5 | 23 | 0 | 23 | Verona | - | Verona | VR | Provincia | 3.096255e+09 | POLYGON ((10.88379 45.77281, 10.88301 45.77202... |
| 23 | 2 | 5 | 24 | 0 | 24 | Vicenza | - | Vicenza | VI | Provincia | 2.720772e+09 | POLYGON ((11.53891 46.01330, 11.54452 46.01176... |
64 rows × 12 columns
fig, ax = plt.subplots(figsize=(10, 10))
target_zones.plot(
ax=ax,
column='COD_REG',
)
plt.title("Regioni interessate dall'analisi sul PM10")
plt.axis('off')
plt.show()
Aggregazione dei dati¶
Lo scopo di questa attività è stato quello di aggregare i dati su base provinciale e di generare un .csv per Provincia. I tempi di calcolo sono davvero lunghi, notate la funzione %%time cosa stampa!
NB. Per poter gestire in velocità il notebook dopo l'aggregazione dei dati ho commentato il codice e fatto uno screenshot al risultato sui tempi di processamento.
csv_folder = Path("/home/max/Desktop/pianura_padana/processed/target_zones")
# %%time
#
# # Analisi per Provincia
# for _index, _row in target_zones.iterrows():
# zone_gdf = target_zones[target_zones['COD_PROV'] == _row['COD_PROV']]
#
# zone_data = []
#
# # Lettura dei singoli `.nc`
# for file in nc_files:
# year_data = xr.open_dataset(
# filename_or_obj=file,
# engine="netcdf4",
# decode_coords="all",
# )
#
# # Riordinamento delle coordinate per evitare problemi di dimensione
# year_data = year_data.transpose('lat', 'lon', 'time')[['lat', 'lon', 'time', 'particulate_matter_10um']]
#
# time_coords = year_data['time'].values
#
# # Calcolo delle zonal statistic
# for time in time_coords:
# sensing_date = time.astype('datetime64[s]').item()
#
# time_data = year_data.sel(time=time).to_dataarray().squeeze()
#
# # Definizione delle dimensioni spaziali
# time_data = time_data.rio.set_spatial_dims(x_dim='lon', y_dim='lat')
#
# # Rasterizzazione del poligono della Provincia
# rasterized_vector = make_geocube(
# vector_data=zone_gdf,
# measurements=['COD_PROV'],
# like=time_data,
# )
# vector_dataarray = rasterized_vector['COD_PROV']
#
# zonal_statistics = stats(
# zones=vector_dataarray,
# values=time_data,
# stats_funcs=["mean", "max", "min", "sum", "std", "count", "var"]
# )
# zonal_statistics.rename(columns={"zone": 'COD_PROV'}, inplace=True)
#
# zs_merged = pd.merge(
# left=zonal_statistics,
# right=zone_gdf,
# how='left',
# on='COD_PROV'
# )
# zs_merged = zs_merged[['COD_PROV', 'DEN_UTS', 'mean', 'max', 'min', 'sum', 'std', 'count', 'var']]
# zs_merged['sensing_date'] = sensing_date
#
# zone_data.append(zs_merged)
# #break
#
#
# #break
# # Aggregazione e generazione del csv
# zone_ddf = dd.concat(zone_data)
# zone_df = zone_ddf.compute()
#
# zone_df.to_csv(csv_folder.joinpath(f"zs_{_row['COD_PROV']}_{_row['DEN_UTS']}.csv"))
#
#
# #break
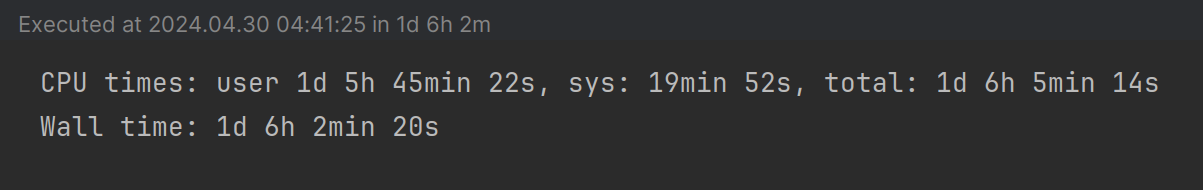
La configurazione dell'hardware che ho usato per questa analisi è quella di un Dell XPS15 9530 con CPU i7-13700H, RAM 64GB DDR5, GPU NVIDIA GeForce RTX4050 con 6GB GDDR6.
A questo punto possiamo creare il DataFrame con cui svilupperemo le nostre analisi usando i quasi 1GB di dati elaborati con le analisi precedenti.
%%time
dask_df = dd.read_csv(str(csv_folder / "*.csv"))
combined_ddf = dask_df.compute()
combined_ddf.drop(columns=['Unnamed: 0'], inplace=True)
combined_ddf['sensing_date'] = pd.to_datetime(combined_ddf['sensing_date'])
combined_ddf = combined_ddf[['DEN_UTS', 'sensing_date', 'min', 'max', 'mean', 'sum']]
combined_ddf.rename(columns={'DEN_UTS': 'Provincia'}, inplace=True)
combined_ddf.set_index(['Provincia', 'sensing_date'], inplace=True)
combined_ddf.sort_index(inplace=True, ascending=True)
combined_ddf
CPU times: user 14.7 s, sys: 3.3 s, total: 18 s Wall time: 6.69 s
| min | max | mean | sum | ||
|---|---|---|---|---|---|
| Provincia | sensing_date | ||||
| Alessandria | 2013-01-01 00:00:00 | 17.774128 | 73.087227 | 60.116840 | 2524.907227 |
| 2013-01-01 01:00:00 | 34.121521 | 77.894402 | 67.446754 | 2832.763672 | |
| 2013-01-01 02:00:00 | 30.085140 | 77.120522 | 65.277397 | 2741.650635 | |
| 2013-01-01 03:00:00 | 28.961617 | 70.821129 | 57.437000 | 2412.354004 | |
| 2013-01-01 04:00:00 | 28.968790 | 60.781708 | 50.913219 | 2138.355225 | |
| ... | ... | ... | ... | ... | ... |
| Vicenza | 2024-03-12 19:00:00 | 6.001117 | 44.228996 | 24.173279 | 725.198364 |
| 2024-03-12 20:00:00 | 5.843284 | 51.150700 | 25.821503 | 774.645081 | |
| 2024-03-12 21:00:00 | 6.246823 | 50.693604 | 25.173981 | 755.219421 | |
| 2024-03-12 22:00:00 | 5.438911 | 56.303368 | 23.828772 | 714.863159 | |
| 2024-03-12 23:00:00 | 4.262040 | 58.259407 | 22.019205 | 660.576172 |
6280704 rows × 4 columns
In questo passaggio mi interessava inserire nel DataFrame finale solo alcuni parametri statistici rispetto a tutti quelli che calcola la funzione di zonal statistics.
Conteggio degli sforamenti del limite normativo¶
Come indicato nell'articolo mi è interessato contare quante volte negli anni c'è stato lo sforamento del limite normativo.
%%time
# Calcolo della media giornaliera
target_stat_count = 'mean'
daily_mean = combined_ddf
daily_mean = daily_mean.groupby(['Provincia', pd.Grouper(level='sensing_date', freq='D')])[target_stat_count].mean()
daily_mean_df = pd.DataFrame(daily_mean)
# Calcolo degli sforamenti
target_column_name = 'over_limit'
count_over_limit = daily_mean_df.loc[daily_mean_df[target_stat_count] >= 50].copy()
count_over_limit[target_column_name] = 1
count_over_limit.drop(columns=[target_stat_count], inplace=True)
count_over_limit = count_over_limit.groupby(['Provincia', pd.Grouper(level='sensing_date', freq='YE', dropna=False)])[target_column_name].sum()
over_limit_df = pd.DataFrame(count_over_limit)
over_limit_df
CPU times: user 808 ms, sys: 239 ms, total: 1.05 s Wall time: 1.05 s
| over_limit | ||
|---|---|---|
| Provincia | sensing_date | |
| Alessandria | 2013-12-31 | 8 |
| 2014-12-31 | 5 | |
| 2015-12-31 | 9 | |
| 2016-12-31 | 13 | |
| 2017-12-31 | 3 | |
| ... | ... | ... |
| Vicenza | 2020-12-31 | 13 |
| 2021-12-31 | 15 | |
| 2022-12-31 | 17 | |
| 2023-12-31 | 27 | |
| 2024-12-31 | 21 |
462 rows × 1 columns
Una volta ottenuto il dato di mio interesse ho opportunamente ridisposto i dati in modo da poterli poi mettere su un grafico.
%%time
pivot_over_limit_df = over_limit_df.pivot_table(index='Provincia', columns='sensing_date', values=target_column_name)
pivot_over_limit_df.fillna(0, inplace=True)
pivot_over_limit_df
CPU times: user 3.29 ms, sys: 736 µs, total: 4.03 ms Wall time: 3.58 ms
| sensing_date | 2013-12-31 | 2014-12-31 | 2015-12-31 | 2016-12-31 | 2017-12-31 | 2018-12-31 | 2019-12-31 | 2020-12-31 | 2021-12-31 | 2022-12-31 | 2023-12-31 | 2024-12-31 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Provincia | ||||||||||||
| Alessandria | 8.0 | 5.0 | 9.0 | 13.0 | 3.0 | 2.0 | 9.0 | 0.0 | 6.0 | 1.0 | 2.0 | 8.0 |
| Ancona | 0.0 | 1.0 | 2.0 | 1.0 | 0.0 | 0.0 | 0.0 | 3.0 | 7.0 | 1.0 | 0.0 | 0.0 |
| Aosta | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 |
| Arezzo | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 3.0 | 3.0 | 1.0 | 0.0 | 0.0 |
| Ascoli Piceno | 0.0 | 4.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 5.0 | 0.0 | 0.0 | 0.0 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| Venezia | 15.0 | 12.0 | 39.0 | 21.0 | 20.0 | 6.0 | 17.0 | 25.0 | 17.0 | 25.0 | 32.0 | 24.0 |
| Verbano-Cusio-Ossola | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 |
| Vercelli | 3.0 | 1.0 | 4.0 | 0.0 | 2.0 | 0.0 | 4.0 | 0.0 | 5.0 | 2.0 | 0.0 | 3.0 |
| Verona | 19.0 | 18.0 | 36.0 | 34.0 | 30.0 | 4.0 | 26.0 | 33.0 | 27.0 | 32.0 | 48.0 | 32.0 |
| Vicenza | 6.0 | 6.0 | 13.0 | 16.0 | 13.0 | 2.0 | 9.0 | 13.0 | 15.0 | 17.0 | 27.0 | 21.0 |
64 rows × 12 columns
fig1, ax1 = plt.subplots(figsize=(10, 20))
fig1.suptitle('Giorni di sforamento di PM10\nal 15-03-2024 a quota 0 metri', fontsize=20)
sns.heatmap(pivot_over_limit_df, cmap="hot", ax=ax1, annot=True)
description = ("\nIl D.Lgs.155/2010 per il PM10 impone un massimo di 35 sforamenti annui del valore limite di\n"
"concentrazione di 50 µg/m3 calcolato su media giornaliera.\n")
ax1.set_title(description, loc='left', fontsize=10)
ax1.set_xlabel('')
ax1.set_ylabel('Province')
ax1.xaxis.tick_top()
labels1= [item.get_text()[:4] for item in ax1.get_xticklabels()]
ax1.set_xticklabels(labels1)
ax1.tick_params(axis='x', rotation=0)
cbar1 = ax1.collections[0].colorbar
cbar1.set_label('Sforamenti')
plt.tight_layout()
plt.show()
Cumulo di PM10 annuale¶
Come accennavo nell'articolo ero curioso di sapere quanta PM10 si accumulava di anno in anno.
%%time
# Calcolo della somma annuale
yearly_mean = combined_ddf
yearly_mean = yearly_mean.groupby(['Provincia', pd.Grouper(level='sensing_date', freq='YE')])['sum'].sum()
yearly_mean_df = pd.DataFrame(yearly_mean)
# Conversione da µg/m3 a g/m3
yearly_mean_df['sum'] *= 0.000001
yearly_mean_df['sum'] = yearly_mean_df['sum'].round(0)
yearly_mean_df
CPU times: user 786 ms, sys: 216 ms, total: 1 s Wall time: 1 s
| sum | ||
|---|---|---|
| Provincia | sensing_date | |
| Alessandria | 2013-12-31 | 8.0 |
| 2014-12-31 | 6.0 | |
| 2015-12-31 | 8.0 | |
| 2016-12-31 | 8.0 | |
| 2017-12-31 | 7.0 | |
| ... | ... | ... |
| Vicenza | 2020-12-31 | 6.0 |
| 2021-12-31 | 6.0 | |
| 2022-12-31 | 6.0 | |
| 2023-12-31 | 6.0 | |
| 2024-12-31 | 2.0 |
768 rows × 1 columns
%%time
pivot_yearly = yearly_mean_df.pivot_table(index='Provincia', columns='sensing_date', values='sum')
pivot_yearly.fillna(0, inplace=True)
pivot_yearly
CPU times: user 3.66 ms, sys: 0 ns, total: 3.66 ms Wall time: 3.11 ms
| sensing_date | 2013-12-31 | 2014-12-31 | 2015-12-31 | 2016-12-31 | 2017-12-31 | 2018-12-31 | 2019-12-31 | 2020-12-31 | 2021-12-31 | 2022-12-31 | 2023-12-31 | 2024-12-31 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Provincia | ||||||||||||
| Alessandria | 8.0 | 6.0 | 8.0 | 8.0 | 7.0 | 7.0 | 7.0 | 6.0 | 6.0 | 7.0 | 6.0 | 2.0 |
| Ancona | 3.0 | 3.0 | 4.0 | 4.0 | 3.0 | 3.0 | 3.0 | 3.0 | 3.0 | 3.0 | 3.0 | 1.0 |
| Aosta | 4.0 | 3.0 | 4.0 | 3.0 | 3.0 | 3.0 | 3.0 | 3.0 | 3.0 | 3.0 | 2.0 | 0.0 |
| Arezzo | 5.0 | 5.0 | 6.0 | 6.0 | 5.0 | 4.0 | 5.0 | 4.0 | 4.0 | 5.0 | 5.0 | 1.0 |
| Ascoli Piceno | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 0.0 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| Venezia | 5.0 | 5.0 | 6.0 | 6.0 | 5.0 | 5.0 | 5.0 | 5.0 | 5.0 | 6.0 | 6.0 | 2.0 |
| Verbano-Cusio-Ossola | 3.0 | 2.0 | 3.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 2.0 | 0.0 |
| Vercelli | 4.0 | 3.0 | 5.0 | 4.0 | 4.0 | 3.0 | 4.0 | 3.0 | 3.0 | 3.0 | 3.0 | 1.0 |
| Verona | 8.0 | 7.0 | 9.0 | 8.0 | 8.0 | 7.0 | 8.0 | 8.0 | 7.0 | 8.0 | 8.0 | 3.0 |
| Vicenza | 6.0 | 5.0 | 7.0 | 7.0 | 6.0 | 5.0 | 6.0 | 6.0 | 6.0 | 6.0 | 6.0 | 2.0 |
64 rows × 12 columns
fig2, ax2 = plt.subplots(figsize=(10, 20))
fig2.suptitle('Cumulo annuo di PM10\nal 15-03-2024 a quota 0 metri\n', fontsize=20)
sns.heatmap(pivot_yearly, cmap="hot", ax=ax2, annot=True)
ax2.set_xlabel('')
ax2.set_ylabel('Province')
ax2.xaxis.tick_top()
labels2 = [item.get_text()[:4] for item in ax2.get_xticklabels()]
ax2.set_xticklabels(labels2)
ax2.tick_params(axis='x', rotation=0)
cbar2 = ax2.collections[0].colorbar
cbar2.set_label('g/m3')
plt.tight_layout()
plt.show()
Conclusione¶
Questo è l'intero procedimento che mi ha portato a mettere insieme i dati per l'articolo sull'andamento del PM10 nel Nord Italia. Una volta aggregati i NetCDF per anno sono riuscito a processare il tutto non senza difficoltà e direi che la maggiore è stata la difficoltà tempo di elaborazione. Devo approfondire meglio Dask per capire se posso evitare di aspettare più di un giorno per la generazione dei .csv per ogni Provincia ed un'altra cosa che vorrei fare è mettere su mappa le aree che sforano i limiti normativi.